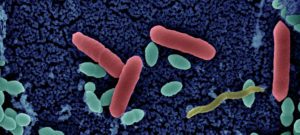
À l’origine de nombreuses maladies infectieuses, les bactéries peuvent également servir de modèle pour étudier des mécanismes biologiques fondamentaux. Les études menées dans le département de Microbiologie portent sur la caractérisation moléculaire des fonctions qui permettent aux bactéries d’interagir avec leur environnement et, pour certaines, de provoquer des maladies. Les scientifiques du département de Microbiologie étudient, au niveau cellulaire et moléculaire, divers micro-organismes (bactéries, archées et leurs virus) en tant que systèmes modèles pour des analyses fondamentales en génomique, génétique, métabolisme, etc. Ils s’intéressent également aux mécanismes qui permettent à certains d’entre eux d’être pathogènes et d’échapper au système immunitaire de l’hôte, ou de résister aux antibiotiques. Pour ces travaux, les chercheurs du département de Microbiologie possèdent une grande variété d’expertises et d’approches expérimentales. Ces études apportent non seulement une meilleure compréhension du mode de vie de ces micro-organismes, mais ils sont également un préalable indispensable au développement de nouvelles thérapies ou de nouveaux outils diagnostiques potentiellement utilisables pour le traitement des infections bactériennes. Le Département de Microbiologie est composé de 21 entités, dont 16 entités de recherche, 1 laboratoire Université de Paris Cité et 4 collections. Trois de ces entités abritent des Centres Nationaux de Référence et 2 unités du Département sont également Centre collaborateur de l’Organisation Mondiale de la Santé (OMS). Par ailleurs, plusieurs unités de recherche d’autres départements (Génomes & Génétique, Biologie Structurale & Chimie et Infection & Epidémiologie) sont affiliées au Département de Microbiologie. Le département héberge également une équipe de recherche labélisée avec le CNRS – UMR6047.
_______________________
Click to access to the
______________________

(2023) Chromosome folding and prophage activation reveal specific genomic architecture for intestinal bacteria., Microbiome May; 11(1): 111.
Bacterial capsular polysaccharides with antibiofilm activity share common biophysical and electrokinetic properties., Nat Commun May; 14(1): 2553.
Elucidating dynamic anaerobe metabolism with HRMAS 13C NMR and genome-scale modeling., Nat Chem Biol May; 19(5): 556-564.
Legionella para-effectors target chromatin and promote bacterial replication., Nat Commun Apr; 14(1): 2154.
Cas9 off-target binding to the promoter of bacterial genes leads to silencing and toxicity. , Nucleic Acids Res Mar; (): .
Yersiniomics, a Multi-Omics Interactive Database for Yersinia Species., Microbiol Spectr Feb; (): e0382622.
Microbiota-induced active translocation of peptidoglycan across the intestinal barrier dictates its within-host dissemination., Proc Natl Acad Sci U S A Jan; 120(4): e2209936120.
Bioenergetic State of Escherichia coli Controls Aminoglycoside Susceptibility., mBio Jan; (): e0330222.
The cnf1 gene is associated with an expanding Escherichia coli ST131 H30Rx/C2 subclade and confers a competitive advantage for gut colonization., Gut Microbes ; 14(1): 2121577.
An ancient divide in outer membrane tethering systems in bacteria suggests a mechanism for the diderm-to-monoderm transition., Nat Microbiol Mar; 7(3): 411-422.
Multilateral benefit-sharing from digital sequence information will support both science and biodiversity conservation., Nat Commun 02; 13(1): 1086.