L’Unité de Spectrométrie de Masse pour la Biologie est, depuis le 1er janvier 2017, une Unité de Service et de Recherche mixte Institut Pasteur/CNRS (USR2000). L’unité intègre un groupe de recherche et la plateforme protéomique de l’Institut Pasteur et fait partie du département de biologie structurale et chimie (DBSC) et du C2RT.
Les activités du groupe de recherche sont focalisées sur le développent d’approches innovantes en protéomique structurale: protéomique top-down, pontage covalent ou échanges H/D couplés à la spectrométrie de masse ou encore spectrométrie de masse native.
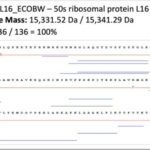
La protéomique top-down est une technologie émergente basée sur l’analyse de protéines intactes, sans digestion enzymatique. Les approches top-down permettent ainsi de caractériser les différentes protéoformes d’une même protéine, liées à la présence de modifications post-traductionnelles, de variants, de polymorphismes simple nucléotide… Elles apportent donc un niveau de précision inégalée au niveau moléculaire, pour les protéines identifiées. La protéomique top-down ouvre des perspectives très intéressantes en recherche clinique, et en particulier en microbiologie clinique, un axe de recherche majeur de l’unité.
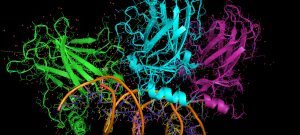
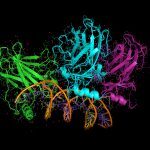
Le pontage covalent combiné à la spectrométrie de masse est une approche puissante permettant de s’intéresser à la structure tridimensionnelle et la topologie de complexes protéiques (surfaces d’interaction entre les sous-unités)… Notre Unité a mis au point récemment un nouvel agent pontant trifonctionnel, basé sur de la chimie click, dont l’utilisation sur des complexes membranaires de très haut poids moléculaire est en cours d’optimisation.
La spectrométrie de masse native permet de s’intéresser à la structure d’édifices supra-moléculaires de très grande taille, avec la vitesse, la précision et la sensibilité de la spectrométrie de masse. Une mesure de masse précise du complexe protéique peut être réalisée, et des informations sur la stoechiométrie, la conformation ou l’arrangement des sous-unités obtenues par des expériences MS ou MS/MS.
Les échanges H/D couplés à la spectrométrie de masse permettent de sonder l’accessibilité au solvant et la présence de liaisons hydrogène dans les différentes zones de la protéine. Les approches HX-MS peuvent ainsi être utilisées pour caractériser des changements de conformation, ou des interactions protéine/protéine. Une des applications majeures de ces approches est l’epitope mapping, pour laquelle le laboratoire a une expertise de longue date.