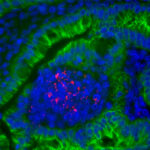
Les modifications de la chromatine, au niveau de l’ADN ou des histones, jouent un rôle fondamental dans la régulation de l’expression des gènes chez les eucaryotes, en contrôlant l’accès de la machinerie transcriptionnelle aux séquences promotrices. Des études récentes ont mis en évidence que modifier la chromatine est l’un des moyens par lesquels les bactéries pathogènes interfèrent avec le programme transcriptionnel des cellules hôtes. Cependant, les mécanismes moléculaires sous-jacents sont très peu caractérisés, et les rôles de ces modifications pour l’hôte ou pour la bactérie restent méconnus. Notre équipe étudie cette facette des interactions hôtes-bactéries en utilisant 2 modèles bactériens, Listeria monocytogenes et Streptococcus pneumoniae, un pathogène et un colonisateur naturel respectivement. Le but étant de déterminer le rôle des modifications de la chromatine induite lors d’interactions hôte bactéries et leurs conséquences à long terme. Nous travaillons à l’interface entre la microbiologie, l’épigénomique et l’immunité innée. Cette approche pluridisciplinaire permettra la découverte de stratégies utilisées par les bactéries afin de reprogrammer la transcription de l’hôte pendant la colonisation et l’infection, et apportera une nouvelle compréhension des mécanismes cellulaires fondamentaux comme la tolérance et la mémoire de l’immunité innée. Par ailleurs, la connaissance des régulations épigénomique induites par les bactéries sur les réponses immunitaires pourrait aboutir au développement de nouveaux traitements antimicrobiens.
Cliquez pour voir le graph
Connexions
Présentation
Membres

Mélanie Hamon
Responsable de Structure
Responsable
Anciens Membres
2000
2000
Name
Position
2016
2016
Paul Primard
Medecine student
2017
2018
Kayley Pate
Master student
2015
2018
Jorge Pereira
PhD student
2018
2019
Emma Patey
Master student
2016
2019
Orhan Rasid
Post-doc
2016
2020
Wenyang Dong
PhD student
2019
2021
Matthew Eldridge
Post-doc
2022
2022
Blaise Hebert
Master student
2022
2022
Fanette Seite
Engineer student
2022
2022
Mateusz Checinski
Master student
2019
2022
Tiphaine Camarasa
PhD student
2023
2023
Aymeric Zellner
Licence student
2023
2023
Thomas Harivel
Research Engineer
2016
2023
Marie-Anne LIN
Administrative Assistant
2024
2024
Endre Csaba PAL
Medecine student
2020
2025
Justine Matheau
PhD student
2023
2025
Anna Both
Physician