Présentation
The Paris Autism Research International Sib-pair (PARIS) Study has been launched as a collaborative project by Prof. M. Leboyer (Paris) and Prof. C. Gillberg (Göteborg) in the 2000s. Its main goal is to join the efforts of clinicians in France and Sweden with the purpose of gathering a large population of individuals with ASD and their first degree relatives to perform genetic studies. All the participants of this study are deep phenotyped. We collect clinical data of all participants. These data include symptoms, comorbidities, quality of life, level of adaptive function, neurocognitive profile and prenatal risk factors. We also collect behavior tests, eye-tracking and data on sensory-motors ability. The clinical data are used to create a comprehensive profile of neurocognitive strengths and weaknesses for each participant.
We also collect brain imaging data (EEG, MRI) for a majority of participants. MRI data are acquired for the majority of the participants at 3T, and include structural T1W data, functional MRI data (up to 3 blocks of 4 min each per participant), and diffusion weighted MRI (32 or 64 directions). For the sMRI, we use automatic and manual segmentation to reconstruct subcortical structures and the neocortical surface. This allows us to measure parameters such as the regional volume, cortical surface area, thickness, grey-white matter contrast or degree of folding. The diffusion weighted MRI data allows us to build whole brain tractography and connectivity matrices. The fMRI data allows us to extract time series for brain activity during cognitive tasks and to construct functional connectivity matrices (resting state). Our EEG protocol includes resting state, standard tasks such as action execution and auditory paradigms (e.g. to study MisMatch Negativity) activity, but also a new paradigm of real-time social interaction with a virtual partner (the Human Dynamic Clamp). Combining high-density EEG helmets with photogrammetry, we can register the electrodes positions in 3D to the anatomical MRI of the participants and then reconstruct electrophysiological activity at the level of cortical and cerebellar sources.
For all participants we collect blood and/or saliva to perform genetic analyses. We are using Illumina SNP arrays and WES/WGS. Structural variants are called using Smoove v0.2.3, Manta v1.6.0, Gridss v2.8.1, QuantiSNP v2.3 and PennCNV v1.0.3, annotated with ANNOVAR or VEP v99 and visualized using IGV v2.4.17, SVPV v1.0, or SnipPeep v1.6. Variants are called using GATK v4.1.4.1 with Best Practices, annotated with VEP v99, and the corresponding CRAM files are visualized using IGV v2.4.17. Deleteriousness prediction is performed using tools such as CADD v1.3. All de novo variants impacting autism/NDD genes are confirmed by Sanger sequencing in our molecular biology lab. We use GRAVITY for visualization of all variants on protein-protein interaction networks. Ancestry estimations are performed using ADMIXTURE v1.3.0, GWAS analyses are conducted using plink v1.9, PCA analyses with fastPCA v1.0.3, polygenic score calculations with PRSice v2.2.13.
All these data put together let us compute genetics, clinical and brain profiles. With the integration of these data, we can stratify the patients more precisely in more homogeneous groups.
All the data from the PARIS project are aligned with the data collected in other research projects like AIMS-2-Trials so that they can be compared and all the analyses can be replicated on different datasets. The data we collect are also compared with data from public databases like UKBiobank, the Psychiatric Genetics Consortium (PGC), ABIDE, ALSPAC or MSSNG.
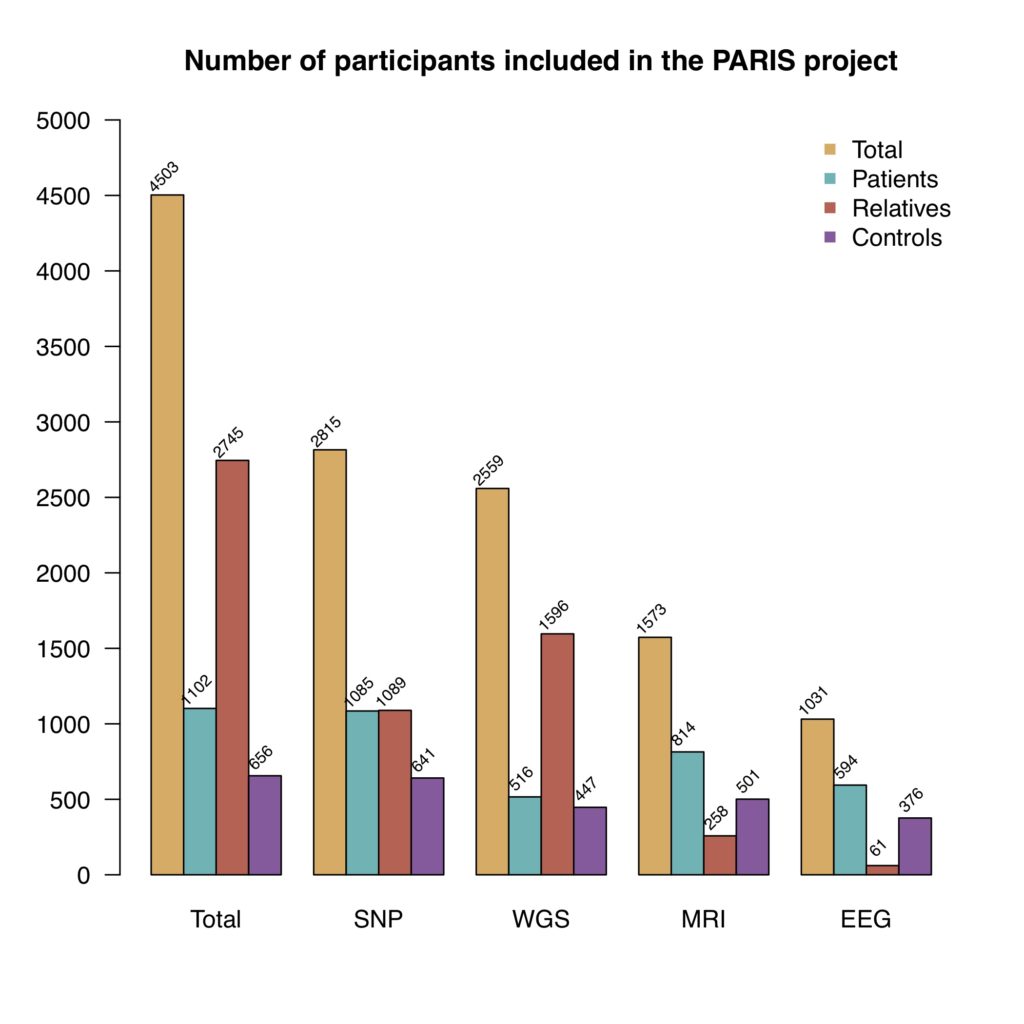
The participants are enrolled in the study at different clinical centers in France and to date, the PARIS cohort consists of 4,503 individuals (1,102 probands with autism, 2,745 relatives and 656 controls). The number of participants for which we have SNP, WGS, MRI and EEG data are presented in the figure below.