Le Centre National de Référence des Escherichia coli, Shigella et Salmonella (CNR-ESS) a été créé par le décret n° 2016-806 du 16 juin 2016 pour assurer la surveillance microbiologique des infections transmissibles suivantes : shigelloses (dysenterie bacillaire), diarrhées sanglantes et syndromes hémolytiques et urémiques causés par les E. coli entérohémorragiques, fièvres typhoïdes et paratyphoïdes et autres salmonelloses (cas sporadiques ou toxi-infections alimentaires collectives).
Le CNR-ESS, situé à l’Institut Pasteur à Paris, gère un réseau national et ultramarin de 1400 laboratoires privés et hospitaliers. A ce CNR est associé le laboratoire de Microbiologie de l’Hôpital Robert-Debré pour la surveillance des syndromes hémolytiques et urémiques grâce à un réseau de pédiatres-néphrologues.
Les missions spécifiques du CNR, telles que définies au cahier des charges de l’appel à candidature (2016) sont :
- Apporter une expertise microbiologique :
- pour le diagnostic des infections à E. coli entérohémorragiques (EHEC) (souches bactériennes, sérologies et mise en évidence des E. coli producteurs de shigatoxines (STEC) dans les selles),
- pour l’identification et le typage des souches de E. coli, shigelles et salmonelles par identification biochimique, sérotypage et/ou moléculaires (PFGE, MLST, MLVA, CRISPR typing et séquençage complet du génome),
- pour la réalisation d’antibiogramme selon l’EUCAST et la caractérisation des mécanismes de résistance aux antibiotiques et de leurs supports génétiques,
- en contribuant au développement de nouvelles méthodes de typage pour tous ces pathogènes,
- en mettant ces méthodes à la disposition des laboratoires intéressés et en contribuant à leur diffusion large,
- en développant l’information et la formation des laboratoires par la diffusion de guides techniques.
- Contribuer à la surveillance épidémiologique, en lien avec Santé publique France (SpF) :
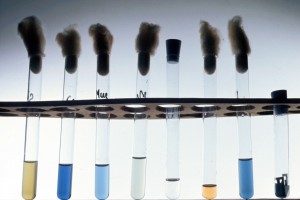
Galerie de sucres mis en présence de bactéries Salmonelles. Fermentation de sucres.
- en transmettant à l’SpF, en temps réel, des résultats concernant les infections à EHEC, à l’investigation des toxi-infections alimentaires collectives à Salmonella et à la déclaration des salmonelloses majeures responsables de fièvres typhoïdes et paratyphoïdes,
- en signalant à Santé publique France les foyers de cas groupés notifiés au CNR, en détectant précocement les épisodes épidémiques, par la caractérisation des souches par la méthode de typage la plus adaptée et par le développement de seuils d’alerte,
- en comparant les souches isolées chez les malades et dans d’autres sources en particulier alimentaire,
- en suivant les tendances évolutives temporelles des différentes espèces de shigelles et de sérotypes de salmonelles, en particulier celles résistantes aux antibiotiques,
- en contribuant aux réseaux de surveillance internationaux et en particulier européens de l’ECDC et de l’EFSA, notamment dans le cadre de l’application de la directive zoonoses 2003/99/CE.
- Contribuer à l’alerte en signalant à Santé publique France tout événement inhabituel : augmentation du nombre de cas, modification des formes cliniques (répartition, modification de leur expression clinique, formes inhabituelles), survenue de cas groupés, apparition de souches inhabituelles, etc.
Intérêt pour la santé publique et la recherche
Un algorithme informatisé de surveillance et d’alerte des infections à Salmonella, issu d’une collaboration CNR-ESS/SpF, permet de détecter précocement au niveau national, régional ou départemental toute augmentation anormale du nombre d’isolements de ces bactéries. Cet algorithme est réévalué de façon régulière par des biostatisticiens.
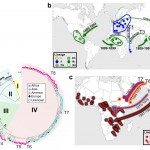
Le CNR-ESS est une entité clé de l’Unité des Bactéries Pathogènes Entériques. L’activité de surveillance et d’expertise du CNR-ESS (tel que le suivi des souches épidémiques ou multirésistantes aux antibiotiques) nourrit les travaux de recherche de l’Unité. Par exemple, la nécessité de pouvoir rapidement « typer », c’est à dire de mettre en évidence si 2 isolats bactériens sont proches génétiquement (pouvant signifier une même origine alimentaire) ou non, va permettre d’étudier la diversité génétique et l’évolution du groupe bactérien en question au niveau de son génome, dans le temps ou dans l’espace. Pour cela le séquençage complet d’une collection de souches historiques et globales des différents agents pathogènes est en cours. L’analyse des données issues de ces séquences génomiques, en collaboration avec la plateforme PIBnet, permettra d’obtenir de nouveaux outils bio-informatiques de surveillance à haut-débit de ces bactéries responsables d’infections alimentaires. Outils pouvant même être utilisés sur le plan international.
Un site dédié aux activités du CNR-ESS et les modalités d’envoi des souches se trouve ici.