An article published by Stéphane Frémont / Chargé de recherche CNRS
Link: https://www.insb.cnrs.fr/fr/cnrsinfo/un-nouvel-acteur-dans-le-bourgeonnement-du-virus-du-sida
Un nouvel acteur dans le bourgeonnement du virus du SIDA
Plus de 40 millions de personnes vivent avec le VIH-1 (Virus de l’Immunodéficience Humain de type 1), responsable du SIDA. Les thérapies existantes ne permettent pas d’éliminer le virus chez les personnes infectées, démontrant l’importance de rechercher de nouvelles cibles thérapeutiques. Dans une étude publiée dans PNAS, des chercheurs ont découvert qu’une enzyme cellulaire appelée MICAL1 est détournée par le virus pour bourgeonner à la surface de la cellule infectée.
Le bourgeonnement du VIH-1 : une étape du cycle viral encore mal comprise
Le VIH-1 est un virus enveloppé, appartenant à la famille des rétrovirus, qui bourgeonne de la membrane plasmique des cellules infectées. Une fois libéré, le virus peut alors infecter de nouvelles cellules et ainsi se propager dans l’organisme. Comparé à d’autres étapes du cycle viral (entrée du virus, rétrotranscription, intégration et réplication du génome viral), les mécanismes moléculaires permettant la sortie des virus une fois assemblés sont moins bien compris. Pour le VIH-1 et les rétrovirus en général, la machinerie cellulaire de scission ESCRT-III permet de couper la membrane déformée par l’assemblage des protéines virales et ainsi de libérer les virus dans le milieu extracellulaire. Cependant, la membrane plasmique de la cellule est fortement associée à un réseau dense et épais de filaments d’actine, appelé le cortex d’actine, et pourrait représenter une barrière physique contrecarrant la sortie des virus. Le rôle de ce réseau d’actine dans le bourgeonnement du virus reste cependant mal compris.
MICAL1, une enzyme qui désassemble l’actine, est détournée par le virus pour bourgeonner à la surface des cellules infectées
La collaboration biologistes cellulaires, virologues et biophysiciens a permis d’identifier un rôle clé de l’enzyme cellulaire MICAL1 et de son activateur Rab35, dans le bourgeonnement du VIH-1. MICAL1 est une enzyme connue pour oxyder directement les filaments d’actine et de provoquer leur dépolymérisation. Cette protéine est impliquée dans de nombreux mécanismes cellulaires qui dépendent d’un désassemblage local de l’actine, comme la cytocinèse (la dernière étape de la division cellulaire), la migration cellulaire ou certains aspects de la biologie des neurones. Aucun rôle n’avait cependant encore été rapporté pour cette protéine dans les infections virales. Cette étude montre que l’inhibition de l’expression de MICAL1 dans des cellules humaines (cellules épithéliales et macrophages) infectées par le VIH-1 réduit la libération des virus qui s’accumulent à la membrane plasmique de la cellule. En présence de MICAL1, les virus bourgeonnent au niveau de zones de la membrane plasmique présentant un niveau faible d’actine corticale. Cependant, en l’absence de MICAL1, les filaments d’actine du cortex ne sont plus correctement désassemblés au niveau des sites de bourgeonnement du virus, ce qui empêche la déformation de la membrane par les protéines virales Gag et le recrutement de la machinerie cellulaire ESCRT-III. Dans ces conditions, les virus produits par la cellule infectée n’arrivent plus à sortir correctement.
L’activité de MICAL1 est auto-inhibée pour éviter une dépolymérisation complète du réseau d’actine de la cellule. Son activité est donc régulée dans le temps et l’espace par des protéines activatrices, dont la GTPase Rab35. Cette étude montre que Rab35 est localisée au niveau des sites du bourgeonnement du VIH-1 et opère avec MICAL1 pour permettre la libération des virus. Cette étude a également permis d’identifier une nouvelle fonction de MICAL1 qui se montre capable, en plus de dépolymériser les filaments d’actine linéaires, de dissocier les branches des réseaux d’actine branchée par le complexe Arp2/3 et présents au cortex cellulaire.
L’oxydation de l’actine par MICAL1 pourrait de façon générale jouer un rôle clé dans le bourgeonnement d’autres virus enveloppés. Ainsi, l’inhibition chimique de l’enzyme MICAL1 pourrait permettre de réduire la production de VIH-1 et d’autres virus enveloppés (comme Ebola, Marbourg et Lassa) qui bourgeonnent à la membrane plasmique par des mécanismes similaires.
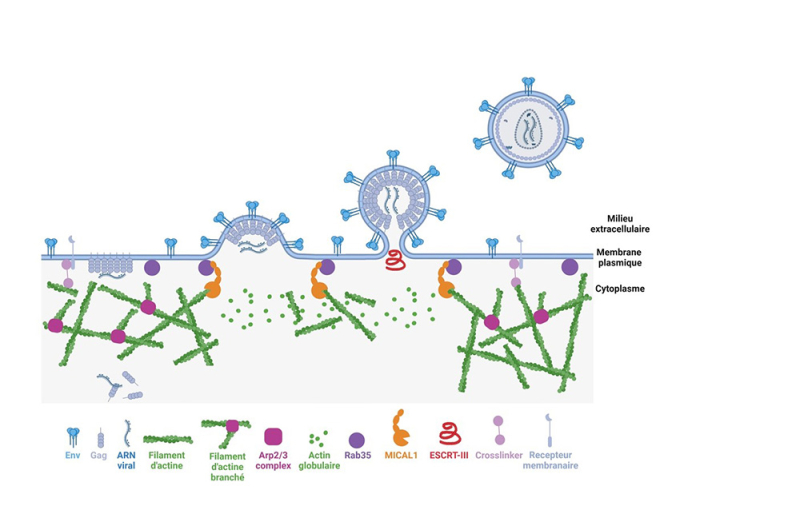
© Thomas Serrano, Stéphane Frémont, Arnaud Echard
Figure : Modèle du rôle de MICAL1 dans le bourgeonnement du VIH-1. La protéine Rab35 est recrutée aux sites de bourgeonnement du VIH-1 où elle active l’enzyme MICAL1. MICAL1 désassemble localement les filaments d’actine, via ses activités de dépolymérisation et de débranchement des filaments, ce qui permet le bourgeonnement du virus et ainsi sa libération par la cellule infectée.
Pour en savoir plus :
HIV-1 budding requires cortical actin disassembly by the oxidoreductase MICAL1
T. Serrano, N. Casartelli, F. Ghasemi, H. Wioland, F. Cuvelier, A. Salles, M. Moya-Nilges, L. Welker, S. Bernacchi, M. Ruff, A. Jégou, G. Romet-Lemonne, O. Schwartz, S. Frémont, A. Echard
18 novembre 2024, DOI: 10.1073/pnas.2407835121